Estudio del mecanismo de iniciación de la traducción del SmRNA del hantavirus andes
2015
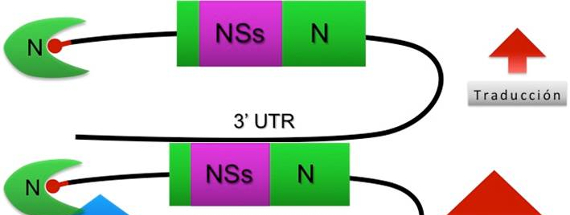
El mRNA del segmento S (SmRNA) del Hantavirus Andes (ANDV) codifica para la proteína multifuncional de la nucleocápside (N). Posee una estructura cap en su extremo 5’ al igual que un mRNA eucarionte, y carece de una cola poli(A) en su extremo 3’. Consistente con estas características, el inicio de la traducción del SmRNA es PABP independiente, y en ausencia de proteínas virales, la traducción del mRNA viral es eIF4G y 3’UTR dependiente.
Nos propusimos caracterizar la participación de N-ANDV en el inicio de la traducción del SmRNA, además de estudiar la participación de proteínas celulares y de la región 3’UTR del SmRNA en la traducción.
Mediante ensayos ex vivo e in vitro, se demostró que la proteína N-ANDV estimula la traducción del SmRNA de manera concentración-dependiente. Adicionalmente, este estudio determinó que N-ANDV es incapaz de reemplazar funcionalmente a eIF4G. Para identificar proteínas celulares que interactúan con la región 3’UTR del SmRNA de ANDV, se usó la técnica “GRNA”. Mediante espectrometría de masas se identificó la proteína hMex-3A, caracterizada como chaperona de RNA. Se evaluó su posible participación en la traducción del SmRNA de ANDV, y se demostró experimentalmente que hMex-3A interactúa con la proteína viral N, estimulando la traducción del mRNA viral.
Con los resultados generados se postuló un modelo de inicio de la traducción del SmRNA, en el cual la proteína N-ANDV es reclutada sobre el extremo 5’ del mensajero, y la proteína hMex-3A se une a la región 3’UTR, incrementando la eficiencia traduccional del SmRNA de ANDV.

- Nombre: Estefanía Castillo Vargas
- Laboratorio: Virología Molecular - Medicina UC
- Mención: Genética Molecular y Microbiología
- Director Tesis: Marcelo López-Lastra
